Keyword: マハラノビス, 二乗距離, 計算, 多変量解析
概要
本サンプルはマハラノビス二乗距離の計算を行うFortranによるサンプルプログラムです。 本サンプルは以下に示されるデータについてマハラノビス二乗距離の計算を行います。
※本サンプルはnAG Fortranライブラリに含まれるルーチン g03dbf() のExampleコードです。本サンプル及びルーチンの詳細情報は g03dbf のマニュアルページをご参照ください。
ご相談やお問い合わせはこちらまで
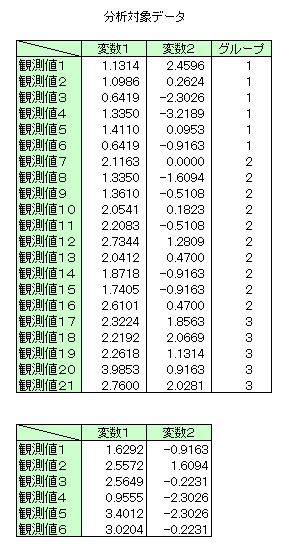
入力データ
(本ルーチンの詳細はg03dbf のマニュアルページを参照)| このデータをダウンロード |
G03DBF Example Program Data 21 2 3 'U' : N,M,NG,WEIGHT 1.1314 2.4596 1 1.0986 0.2624 1 0.6419 -2.3026 1 1.3350 -3.2189 1 1.4110 0.0953 1 0.6419 -0.9163 1 2.1163 0.0000 2 1.3350 -1.6094 2 1.3610 -0.5108 2 2.0541 0.1823 2 2.2083 -0.5108 2 2.7344 1.2809 2 2.0412 0.4700 2 1.8718 -0.9163 2 1.7405 -0.9163 2 2.6101 0.4700 2 2.3224 1.8563 3 2.2192 2.0669 3 2.2618 1.1314 3 3.9853 0.9163 3 2.7600 2.0281 3 : End of X,ING (G03EAF) 1 1 : ISX 'S' 'U' : MODE,EQUAL 6 : NOBS 1.6292 -0.9163 2.5572 1.6094 2.5649 -0.2231 0.9555 -2.3026 3.4012 -2.3026 3.0204 -0.2231 : End of X
- 1行目はタイトル行で読み飛ばされます。
- 2行目に観測値の数(n=21)、変数の数(m=2)、グループの数(ng=3)、重みづけをするかどうか(weight='U':重みづけをしない)を指定しています。
- 3〜23行目に変数の観測値(x)と観測値がどのグループに属するか(ing)を指定しています。
- 24行目に変数が距離計算に含まれるかどうか(isx)を指定しています。"1"の場合は計算に含まれます。
- 25行目に標本点からの距離が計算されるかグループ平均間の距離が計算されるか(mode='S':標本点とグループ平均との間の距離を計算)、グループ内の分散共分散行列が等しいと仮定されるかどうか(equal='U':等しくない)を指定しています。
- 26行目に距離が計算される標本点の数(nobs)を指定しています。
- 27〜32行目に変数の新たな観測値(x)を指定しています。
出力結果
(本ルーチンの詳細はg03dbf のマニュアルページを参照)| この出力例をダウンロード |
G03DBF Example Program Results
Distances
1 2 3
1 3.3393 0.7521 50.9283
2 20.7771 5.6559 0.0597
3 21.3631 4.8411 19.4978
4 0.7184 6.2803 124.7323
5 55.0003 88.8604 71.7852
6 36.1703 15.7849 15.7489
- 4〜10行目に各観測値についてグループ平均からのマハラノビス二乗距離が出力されています。観測値のうち最初の4つはグループのうちの一つに近く、残りの2つはどのグループからも距離があることがわかります。
ソースコード
(本ルーチンの詳細はg03dbf のマニュアルページを参照)
※本サンプルソースコードは科学技術・統計計算ライブラリである「nAG Fortranライブラリ」のルーチンを呼び出します。
サンプルのコンパイル及び実行方法
| このソースコードをダウンロード |
PROGRAM g03dbfe
! G03DBF Example Program Text
! Mark 23 Release. nAG Copyright 2011.
! .. Use Statements ..
USE nag_library, ONLY : g03daf, g03dbf, nag_wp, x04caf
! .. Implicit None Statement ..
IMPLICIT NONE
! .. Parameters ..
INTEGER, PARAMETER :: nin = 5, nout = 6
! .. Local Scalars ..
REAL (KIND=nag_wp) :: df, sig, stat
INTEGER :: i, ifail, ldd, ldgmn, ldox, ldx, &
lgc, lwk, lwt, m, n, ng, nobs, nvar
CHARACTER (1) :: equal, mode, weight
! .. Local Arrays ..
REAL (KIND=nag_wp), ALLOCATABLE :: d(:,:), det(:), gc(:), gmn(:,:), &
ox(:,:), wk(:), wt(:), x(:,:)
INTEGER, ALLOCATABLE :: ing(:), isx(:), iwk(:), nig(:)
! .. Intrinsic Functions ..
INTRINSIC count, max
! .. Executable Statements ..
WRITE (nout,*) 'G03DBF Example Program Results'
WRITE (nout,*)
FLUSH (nout)
! Skip headings in data file
READ (nin,*)
! Read in the problem size
READ (nin,*) n, m, ng, weight
IF (weight=='W' .OR. weight=='w') THEN
lwt = n
ELSE
lwt = 0
END IF
ldox = n
ALLOCATE (ox(ldox,m),ing(n),wt(lwt),isx(m))
! Read in original data
IF (lwt>0) THEN
READ (nin,*) (ox(i,1:m),ing(i),wt(i),i=1,n)
ELSE
READ (nin,*) (ox(i,1:m),ing(i),i=1,n)
END IF
! Read in variable inclusion flags
READ (nin,*) isx(1:m)
! Calculate NVAR
nvar = count(isx(1:m)==1)
ldgmn = ng
lgc = (ng+1)*nvar*(nvar+1)/2
lwk = max(n*(nvar+1),2*nvar)
ALLOCATE (nig(ng),gmn(ldgmn,nvar),det(ng),gc(lgc),wk(lwk),iwk(ng))
! Compute covariance matrix
ifail = 0
CALL g03daf(weight,n,m,ox,ldox,isx,nvar,ing,ng,wt,nig,gmn,ldgmn,det,gc, &
stat,df,sig,wk,iwk,ifail)
! Read in size data from which to compute distances
READ (nin,*) mode, equal
IF (mode=='S' .OR. mode=='s') THEN
READ (nin,*) nobs
ldd = nobs
ELSE
nobs = 0
ldd = ng
END IF
ldx = nobs
ALLOCATE (x(ldx,m),d(ldd,ng))
! Read in data from which to compute distances
IF (nobs>0) THEN
READ (nin,*) (x(i,1:m),i=1,nobs)
END IF
! Compute distances
ifail = 0
CALL g03dbf(equal,mode,nvar,ng,gmn,ldgmn,gc,nobs,m,isx,x,ldx,d,ldd,wk, &
ifail)
! Display results
ifail = 0
CALL x04caf('General',' ',nobs,ng,d,ldd,'Distances',ifail)
END PROGRAM g03dbfe
