Keyword: 制限つき最尤法, REML, 線形混合効果回帰
概要
本サンプルは制限つき最尤法(REML)を用いた線形混合効果回帰のフィットを行うC言語によるサンプルプログラムです。 本サンプルは以下に示されるデータについて線形混合効果回帰のフィットを行います。
※本サンプルはnAG Cライブラリに含まれる関数 nag_reml_mixed_regsn() のExampleコードです。本サンプル及び関数の詳細情報は nag_reml_mixed_regsn のマニュアルページをご参照ください。
ご相談やお問い合わせはこちらまで
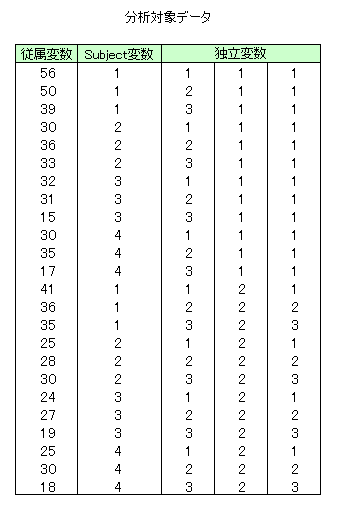
入力データ
(本関数の詳細はnag_reml_mixed_regsn のマニュアルページを参照)| このデータをダウンロード |
nag_reml_mixed_regsn (g02jac) Example Program Data 24 5 3 1 1 1 4 3 2 3 1 3 4 5 3 2 0 1 1 1 56 1 1 1 1 50 1 2 1 1 39 1 3 1 1 30 2 1 1 1 36 2 2 1 1 33 2 3 1 1 32 3 1 1 1 31 3 2 1 1 15 3 3 1 1 30 4 1 1 1 35 4 2 1 1 17 4 3 1 1 41 1 1 2 1 36 1 2 2 2 35 1 3 2 3 25 2 1 2 1 28 2 2 2 2 30 2 3 2 3 24 3 1 2 1 27 3 2 2 2 19 3 3 2 3 25 4 1 2 1 30 4 2 2 2 18 4 3 2 3 1.0 1.0 -1
- 1行目はタイトル行で読み飛ばされます。
- 2行目は観測値の数(n)、データ行列のカラム数(ncol)、固定として処理される独立変数の数(nfv)、ランダムとして処理される独立変数の数(nrv)、分散成分の数(nvpr)を指定しています。
- 3行目は各変数のレベル数(levels)を指定しています。
- 4行目は従属変数をもつデータ行列DATのカラム(yvid)、固定独立変数をもつDATのカラム(fvid)、ランダム独立変数をもつDATのカラム(rvid)、subject(個体)変数をもつDATのカラム(svid)、case weightをもつDATのカラム(cwid)、固定切片が含まれるかを示すフラグ(fint)、ランダム切片が含まれるかどうかを示すフラグ(rint)を指定しています。
- 5行目はランダム変数の分散を示すフラグ(vpr)を指定しています。
- 6〜29行目はデータ行列(dat)を指定しています。
- 30行目はガンマの初期値(gamma)を指定しています
- 31行目は反復の最大値(maxit)を指定しています。ゼロより小さい場合は100が使用されます。
出力結果
(本関数の詳細はnag_reml_mixed_regsn のマニュアルページを参照)| この出力例をダウンロード |
nag_reml_mixed_regsn (g02jac) Example Program Results Fixed effects (Estimate and Standard Deviation) Intercept 37.0000 4.6674 Variable 1 Level 2 1.0000 3.5173 Variable 1 Level 3 -11.0000 3.5173 Variable 2 Level 2 -8.2500 2.1635 Variable 3 Level 2 0.5000 3.0596 Variable 3 Level 3 7.7500 3.0596 Random Effects (Estimate and Standard Deviation) Intercept for Subject Level 1 10.7631 4.4865 Subject Level 1 Variable 1 Level 1 3.7276 3.0331 Subject Level 1 Variable 1 Level 2 -1.4476 3.0331 Subject Level 1 Variable 1 Level 3 0.3733 3.0331 Intercept for Subject Level 2 -0.5269 4.4865 Subject Level 2 Variable 1 Level 1 -3.7171 3.0331 Subject Level 2 Variable 1 Level 2 -1.2253 3.0331 Subject Level 2 Variable 1 Level 3 4.8125 3.0331 Intercept for Subject Level 3 -5.6450 4.4865 Subject Level 3 Variable 1 Level 1 0.5903 3.0331 Subject Level 3 Variable 1 Level 2 0.3987 3.0331 Subject Level 3 Variable 1 Level 3 -2.3806 3.0331 Intercept for Subject Level 4 -4.5912 4.4865 Subject Level 4 Variable 1 Level 1 -0.6009 3.0331 Subject Level 4 Variable 1 Level 2 2.2742 3.0331 Subject Level 4 Variable 1 Level 3 -2.8052 3.0331 Variance Components 1 62.3958 2 15.3819 SIGMA^2 = 9.3611 -2LOG LIKE = 119.7618 DF = 16
- 3〜10行目に固定効果が出力されています。
- 5行目に固定切片と標準誤差が出力されています。
- 6〜10行目に固定独立変数のレベルごとにパラメータ推定と標準偏差が出力されています。
- 12〜28行目にランダム効果が出力されています。
- 13行目にsubject変数のレベル1のランダム切片と標準誤差が出力されています。
- 14〜16行目にランダム独立変数のレベルごとにパラメータ推定と標準偏差が出力されています。
- 17行目にsubject変数のレベル2のランダム切片と標準誤差が出力されています。
- 18〜20行目にランダム独立変数のレベルごとにパラメータ推定と標準偏差が出力されています。
- 21行目にsubject変数のレベル3のランダム切片と標準誤差が出力されています。
- 22〜24行目にランダム独立変数のレベルごとにパラメータ推定と標準偏差が出力されています。
- 25行目にsubject変数のレベル4のランダム切片と標準誤差が出力されています。
- 26〜28行目にランダム独立変数のレベルごとにパラメータ推定と標準偏差が出力されています。
- 30〜32行目に分散成分が出力されています。
- 33行目にシグマの2乗が出力されています。
- 35行目に対数尤度(log-likelihood)が出力されています。
- 37行目に自由度が出力されています。
ソースコード
(本関数の詳細はnag_reml_mixed_regsn のマニュアルページを参照)
※本サンプルソースコードはnAG数値計算ライブラリ(Windows, Linux, MAC等に対応)の関数を呼び出します。
サンプルのコンパイル及び実行方法
| このソースコードをダウンロード |
/* nag_reml_mixed_regsn (g02jac) Example Program.
*
* CLL6I261D/CLL6I261DL Version.
*
* Copyright 2017 Numerical Algorithms Group.
*
* Mark 26.1, 2017.
*/
#include <stdio.h>
#include <nag.h>
#include <nag_stdlib.h>
#include <nagg02.h>
int main(void)
{
/* Scalars */
double like, tol;
Integer cwid, df, exit_status, fint, i, j, k, l, lb, maxit, n, ncol, nff;
Integer nfv, nrf, nrv, nvpr, tddat, rint, svid, warnp, yvid, fnlevel;
Integer rnlevel, lgamma, fl;
/* Nag types */
NagError fail;
/* Arrays */
double *b = 0, *dat = 0, *gamma = 0, *se = 0;
Integer *fvid = 0, *levels = 0, *rvid = 0, *vpr = 0;
#define DAT(I, J) dat[(I-1)*tddat + J - 1]
exit_status = 0;
INIT_FAIL(fail);
printf("nag_reml_mixed_regsn (g02jac) Example Program Results\n\n");
/* Skip heading in data file */
scanf("%*[^\n] ");
/* Read in the problem size information */
scanf("%ld%ld%ld%ld%ld"
"%*[^\n] ", &n, &ncol, &nfv, &nrv, &nvpr);
/* Check problem size */
if (n < 0 || ncol < 0 || nfv < 0 || nrv < 0 || nvpr < 0) {
printf("Invalid problem size, at least one of n, ncol, nfv, "
"nrv or nvpr is < 0\n");
exit_status = 1;
goto END;
}
/* Allocate memory first lot of memory */
if (!(levels = nAG_ALLOC(ncol, Integer)) ||
!(fvid = nAG_ALLOC(nfv, Integer)) ||
!(rvid = nAG_ALLOC(nrv, Integer)) || !(vpr = nAG_ALLOC(nrv, Integer)))
{
printf("Allocation failure\n");
exit_status = -1;
goto END;
}
/* Read in number of levels for each variable */
for (i = 1; i <= ncol; ++i) {
scanf("%ld", &levels[i - 1]);
}
scanf("%*[^\n] ");
/* Read in model information */
scanf("%ld", &yvid);
for (i = 1; i <= nfv; ++i) {
scanf("%ld", &fvid[i - 1]);
}
for (i = 1; i <= nrv; i++) {
scanf("%ld", &rvid[i - 1]);
}
scanf("%ld%ld%ld%ld%*[^\n] ", &svid,
&cwid, &fint, &rint);
scanf("%*[^\n] ");
/* Read in the variance component flag */
for (i = 1; i <= nrv; ++i) {
scanf("%ld", &vpr[i - 1]);
}
scanf("%*[^\n] ");
/* If no subject specified, then ignore rint */
if (svid == 0) {
rint = 0;
}
/* Count the number of levels in the fixed parameters */
for (i = 1, fnlevel = 0; i <= nfv; ++i) {
fl = levels[fvid[i - 1] - 1] - 1;
fnlevel += (fl < 1) ? 1 : fl;
}
if (fint == 1) {
fnlevel++;
}
/* Count the number of levels in the random parameters */
for (i = 1, rnlevel = 0; i <= nrv; ++i) {
rnlevel += levels[rvid[i - 1] - 1];
}
if (rint) {
rnlevel++;
}
/* Calculate the sizes of the output arrays */
if (rint == 1) {
lgamma = nvpr + 2;
}
else {
lgamma = nvpr + 1;
}
if (svid) {
lb = fnlevel + levels[svid - 1] * rnlevel;
}
else {
lb = fnlevel + rnlevel;
}
tddat = ncol;
/* Allocate remaining memory */
if (!(dat = nAG_ALLOC(n * ncol, double)) ||
!(gamma = nAG_ALLOC(lgamma, double)) ||
!(b = nAG_ALLOC(lb, double)) || !(se = nAG_ALLOC(lb, double)))
{
printf("Allocation failure\n");
exit_status = -1;
goto END;
}
/* Read in the Data matrix */
for (i = 1; i <= n; ++i) {
for (j = 1; j <= ncol; ++j) {
scanf("%lf", &DAT(i, j));
}
}
/* Read in the initial values for GAMMA */
for (i = 1; i < lgamma; ++i) {
scanf("%lf", &gamma[i - 1]);
}
/* Read in the maximum number of iterations */
scanf("%ld%*[^\n] ", &maxit);
/* Run the analysis */
tol = 0.;
warnp = 0;
/* nag_reml_mixed_regsn (g02jac).
* Linear mixed effects regression using Restricted Maximum
* Likelihood (REML)
*/
nag_reml_mixed_regsn(n, ncol, dat, tddat, levels, yvid, cwid, nfv, fvid,
fint, nrv, rvid, nvpr, vpr, rint, svid, gamma, &nff,
&nrf, &df, &like, lb, b, se, maxit, tol, &warnp,
&fail);
/* Report the results */
if (fail.code == NE_NOERROR) {
/* Output results */
if (warnp != 0) {
printf("Warning: At least one variance component was ");
printf("estimated to be negative and then reset to zero");
printf("\n");
}
printf("Fixed effects (Estimate and Standard Deviation)\n\n");
k = 1;
if (fint == 1) {
printf("Intercept %10.4f%10.4f\n", b[k - 1], se[k - 1]);
++k;
}
for (i = 1; i <= nfv; ++i) {
for (j = 1; j <= levels[fvid[i - 1] - 1]; ++j) {
if (levels[fvid[i - 1] - 1] != 1 && j == 1)
continue;
printf("Variable%4ld Level%4ld%10.4f%10.4f\n",
i, j, b[k - 1], se[k - 1]);
++k;
}
}
printf("\n");
printf("Random Effects (Estimate and Standard Deviation)\n");
if (svid == 0) {
for (i = 1; i <= nrv; ++i) {
for (j = 1; j <= levels[rvid[i - 1] - 1]; ++j) {
printf("%s%4ld%s%4ld%10.4f%10.4f\n",
"Variable", i, " Level", j, b[k - 1], se[k - 1]);
++k;
}
}
}
else {
for (l = 1; l <= levels[svid - 1]; ++l) {
if (rint == 1) {
printf("%s%4ld%s%10.4f%10.4f\n",
"Intercept for Subject Level", l, " ",
b[k - 1], se[k - 1]);
++k;
}
for (i = 1; i <= nrv; ++i) {
for (j = 1; j <= levels[rvid[i - 1] - 1]; ++j) {
printf("%s%4ld%s%4ld%s%4ld"
"%10.4f%10.4f\n", "Subject Level", l,
" Variable", i, " Level", j, b[k - 1], se[k - 1]);
++k;
}
}
}
}
printf("\n");
printf("%s\n", " Variance Components");
for (i = 1; i <= nvpr + rint; ++i) {
printf("%4ld%10.4f\n", i, gamma[i - 1]);
}
printf("%s%10.4f\n\n", "SIGMA^2 = ", gamma[nvpr + rint]);
printf("%s%10.4f\n\n", "-2LOG LIKE = ", like);
printf("%s%ld\n", "DF = ", df);
}
else {
printf("Routine nag_reml_mixed_regsn (g02jac) failed, with error "
"message:\n%s\n", fail.message);
}
END:
nAG_FREE(b);
nAG_FREE(dat);
nAG_FREE(gamma);
nAG_FREE(se);
nAG_FREE(fvid);
nAG_FREE(levels);
nAG_FREE(rvid);
nAG_FREE(vpr);
return exit_status;
}
