Keyword: 階層的クラスター分析, 多変量解析
概要
本サンプルは階層的クラスター分析を行うC言語によるサンプルプログラムです。 本サンプルは以下に示されるオブジェクトについて階層的クラスター分析を行い、デンドログラム(樹状図)を出力します。
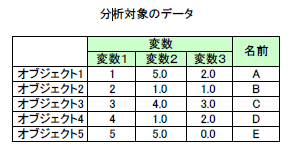
※本サンプルはnAG Cライブラリに含まれる関数 nag_mv_hierar_cluster_analysis() のExampleコードです。本サンプル及び関数の詳細情報は nag_mv_hierar_cluster_analysis のマニュアルページをご参照ください。
ご相談やお問い合わせはこちらまで
入力データ
(本関数の詳細はnag_mv_hierar_cluster_analysis のマニュアルページを参照)| このデータをダウンロード |
nag_mv_hierar_cluster_analysis (g03ecc) Example Program Data 5 3 Nag_Median Nag_NoMatUp Nag_DistSquared Nag_NoVarScale 1 5.0 2.0 A 2 1.0 1.0 B 3 4.0 3.0 C 4 1.0 2.0 D 5 5.0 0.0 E 0 1 1 1.0 1.0 1.0
- 1行目はタイトル行で読み飛ばされます。
- 2行目にオブジェクトの数(n)と変数の総数(m)を指定しています。
- 3行目にクラスタリングの手法(method)を指定しています。"Nag_Median"はメディアン法を意味しています。
- 4行目には既存の距離行列がアップデートされるかを示すパラメータ(update)、どの種類の距離が計算されるかを示すパラメータ(dist)、変数の標準化を示すパラメータ(scale)を指定しています。
この場合、"Nag_NoMatUp" は距離が距離行列に追加される前に行列がゼロに初期化されることを意味しています。 "Nag_DistSquared" はユークリッド平方距離が計算されることを意味しています。"Nag_NoVarScale" は変数がスケーリングされないことを意味しています。 - 5〜9行目にオブジェクトに対する変数の値(x)と名前(name)を指定しています。
- 10行目に変数が距離計算に含まれるかどうかのフラグ(isx)を指定しています。
- 11行目には変数のスケーリングを示すパラメータ(s)を指定しています。スケーリングがないので1.0を指定しています。
出力結果
(本関数の詳細はnag_mv_hierar_cluster_analysis のマニュアルページを参照)| この出力例をダウンロード |
nag_mv_hierar_cluster_analysis (g03ecc) Example Program Results
Distance Clusters Joined
1.000 B D
2.000 A C
6.500 A E
14.125 A B
Dendrogram
14.125 -------
I I
I I
12.006 I I
I I
I I
9.887 I I
I I
I I
7.769 I I
---* I
I I I
5.650 I I I
I I I
I I I
3.531 I I I
I I I
---* I I
1.412 I I I ---*
I I I I I
A C E B D
- 6〜9行目にクラスターの距離と結合されたクラスターが出力されています。
- 11〜34行目にはデンドログラム(樹状図)が出力されています。
ソースコード
(本関数の詳細はnag_mv_hierar_cluster_analysis のマニュアルページを参照)
※本サンプルソースコードはnAG数値計算ライブラリ(Windows, Linux, MAC等に対応)の関数を呼び出します。
サンプルのコンパイル及び実行方法
| このソースコードをダウンロード |
/* nag_mv_hierar_cluster_analysis (g03ecc) Example Program.
*
* CLL6I261D/CLL6I261DL Version.
*
* Copyright 2017 Numerical Algorithms Group.
*
* Mark 26.1, 2017.
*
*/
#include <nag.h>
#include <stdio.h>
#include <nag_stdlib.h>
#include <nagg03.h>
#define X(I, J) x[(I) *tdx + J]
int main(void)
{
Integer exit_status = 0, i, j, m, n, nsym, tdx;
Integer *ilc = 0, *iord = 0, *isx = 0, *iuc = 0;
char **c = 0, name[40][2];
double dmin_, dstep, ydist;
double *cd = 0, *d = 0, *dord = 0, *s = 0, *x = 0;
char nag_enum_arg[40];
Nag_ClusterMethod method;
Nag_DistanceType dist;
Nag_MatUpdate update;
Nag_VarScaleType scale;
NagError fail;
INIT_FAIL(fail);
printf("nag_mv_hierar_cluster_analysis (g03ecc) Example Program "
"Results\n\n");
/* Skip heading in data file */
scanf("%*[^\n]");
scanf("%ld", &n);
scanf("%ld", &m);
if (n >= 2 && m >= 1) {
if (!(cd = nAG_ALLOC(n - 1, double)) ||
!(d = nAG_ALLOC(n * (n - 1) / 2, double)) ||
!(dord = nAG_ALLOC(n, double)) ||
!(s = nAG_ALLOC(m, double)) ||
!(x = nAG_ALLOC(n * m, double)) ||
!(ilc = nAG_ALLOC(n - 1, Integer)) ||
!(iord = nAG_ALLOC(n, Integer)) ||
!(isx = nAG_ALLOC(m, Integer)) || !(iuc = nAG_ALLOC(n - 1, Integer)))
{
printf("Allocation failure\n");
exit_status = -1;
goto END;
}
tdx = m;
}
else {
printf("Invalid n or m.\n");
exit_status = 1;
return exit_status;
}
scanf("%39s", nag_enum_arg);
/* nag_enum_name_to_value (x04nac).
* Converts nAG enum member name to value
*/
method = (Nag_ClusterMethod) nag_enum_name_to_value(nag_enum_arg);
scanf("%39s", nag_enum_arg);
update = (Nag_MatUpdate) nag_enum_name_to_value(nag_enum_arg);
scanf("%39s", nag_enum_arg);
dist = (Nag_DistanceType) nag_enum_name_to_value(nag_enum_arg);
scanf("%39s", nag_enum_arg);
scale = (Nag_VarScaleType) nag_enum_name_to_value(nag_enum_arg);
for (j = 0; j < n; ++j) {
for (i = 0; i < m; ++i)
scanf("%lf", &X(j, i));
scanf("%1s", name[j]);
}
for (i = 0; i < m; ++i)
scanf("%ld", &isx[i]);
for (i = 0; i < m; ++i)
scanf("%lf", &s[i]);
/* Compute the distance matrix */
/* nag_mv_distance_mat (g03eac).
* Compute distance (dissimilarity) matrix
*/
nag_mv_distance_mat(update, dist, scale, n, m, x, tdx, isx, s, d, &fail);
if (fail.code != NE_NOERROR) {
printf("Error from nag_mv_distance_mat (g03eac).\n%s\n", fail.message);
exit_status = 1;
goto END;
}
/* Perform clustering */
/* nag_mv_hierar_cluster_analysis (g03ecc).
* Hierarchical cluster analysis
*/
nag_mv_hierar_cluster_analysis(method, n, d, ilc, iuc, cd, iord, dord,
&fail);
if (fail.code != NE_NOERROR) {
printf("Error from nag_mv_hierar_cluster_analysis (g03ecc).\n%s\n",
fail.message);
exit_status = 1;
goto END;
}
printf("\n Distance Clusters Joined\n\n");
for (i = 1; i <= n - 1; ++i)
printf("%10.3f %3s%3s\n", cd[i - 1], name[ilc[i - 1] - 1],
name[iuc[i - 1] - 1]);
/* Produce dendrogram */
nsym = 20;
dmin_ = 0.0;
dstep = cd[n - 2] / (double) nsym;
/* nag_mv_dendrogram (g03ehc).
* Construct dendrogram following
* nag_mv_hierar_cluster_analysis (g03ecc)
*/
nag_mv_dendrogram(Nag_DendSouth, n, dord, dmin_, dstep, nsym, &c, &fail);
if (fail.code != NE_NOERROR) {
printf("Error from nag_mv_dendrogram (g03ehc).\n%s\n", fail.message);
exit_status = 1;
goto END;
}
printf("\n");
printf("Dendrogram ");
printf("\n");
printf("\n");
ydist = cd[n - 2];
for (i = 0; i < nsym; ++i) {
if ((i + 1) % 3 == 1) {
printf("%10.3f%6s", ydist, "");
printf("%s", c[i]);
printf("\n");
}
else {
printf("%16s%s", "", c[i]);
printf("\n");
}
ydist -= dstep;
}
printf("\n");
printf("%14s", "");
for (i = 0; i < n; ++i) {
printf("%3s", name[iord[i] - 1]);
}
printf("\n");
/* nag_mv_dend_free (g03xzc).
* Frees memory allocated to the dendrogram array in
* nag_mv_dendrogram (g03ehc)
*/
nag_mv_dend_free(&c);
END:
nAG_FREE(cd);
nAG_FREE(d);
nAG_FREE(dord);
nAG_FREE(s);
nAG_FREE(x);
nAG_FREE(ilc);
nAG_FREE(iord);
nAG_FREE(isx);
nAG_FREE(iuc);
return exit_status;
}
