Keyword: クラスター指示変数, 計算, 多変量解析
概要
本サンプルはクラスター指示変数の計算を行うC言語によるサンプルプログラムです。 本サンプルは以下に示されるデータについてクラスター指示変数の計算を行います。
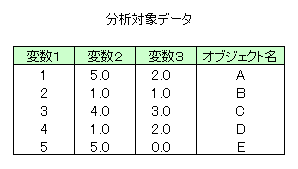
※本サンプルはnAG Cライブラリに含まれる関数 nag_mv_cluster_indicator() のExampleコードです。本サンプル及び関数の詳細情報は nag_mv_cluster_indicator のマニュアルページをご参照ください。
ご相談やお問い合わせはこちらまで
入力データ
(本関数の詳細はnag_mv_cluster_indicator のマニュアルページを参照)| このデータをダウンロード |
nag_mv_cluster_indicator (g03ejc) Example Program Data 5 3 Nag_Median Nag_NoMatUp Nag_DistSquared Nag_NoVarScale 1 5.0 2.0 A 2 1.0 1.0 B 3 4.0 3.0 C 4 1.0 2.0 D 5 5.0 0.0 E 0 1 1 1.0 1.0 1.0 2 0.0
- 1行目はタイトル行で読み飛ばされます。
- 2行目に観測値の数(n)と変数の数(m)を指定しています。
- 3行目にクラスタリングの手法(method)を指定しています。"Nag_Median"はメディアン法(Median)を使用することを意味します。
- 4行目には既存の行列が更新されるかどうかを示すパラメータ(update)、計算される距離の種類(dist)、使用される変数の標準化(scale)を指定しています。この場合、"Nag_NoMatUp"は距離が行列Dに追加される前に行列Dは初期化されることを意味しています。 "Nag_DistSquared" はユークリッド平方距離を意味しています。 "Nag_NoVarScale" はスケールしないことを意味しています。
- 5〜9行目に変数の値(x)とオブジェクト名(name)を指定しています。
- 10行目に変数がクラスター指示変数の計算に含まれるどうかを示すフラグ(isx)を指定しています。"1"の場合は計算に含まれます。
- 11行目は変数に対するスケーリング(s)を指定しますが、"Nag_Nag_NoVarScale"の場合は各変数に対し 1.0 を指定しています。
- 12行目に特定の数のクラスタ−が必要かどうかその数(k)とクラスターが生成される際の距離(dlevel)を指定しています。
出力結果
(本関数の詳細はnag_mv_cluster_indicator のマニュアルページを参照)| この出力例をダウンロード |
nag_mv_cluster_indicator (g03ejc) Example Program Results
Distance Clusters Joined
1.000 B D
2.000 A C
6.500 A E
14.125 A B
Dendrogram
14.125 -------
I I
I I
12.006 I I
I I
I I
9.887 I I
I I
I I
7.769 I I
---* I
I I I
5.650 I I I
I I I
I I I
3.531 I I I
I I I
---* I I
1.412 I I I ---*
I I I I I
A C E B D
Allocation to 2 clusters
Object Cluster
A 1
B 2
C 1
D 2
E 1
- 4〜9行目に距離と結合されたクラスターが出力されています。
- 11〜34行目にデンドログラム(樹状図)が出力されています。
- 36行目に2つのクラスターに割り当てられたことが出力されています。
- 38〜44行目にオブジェクトがどのクラスターに属するかが出力されています。
ソースコード
(本関数の詳細はnag_mv_cluster_indicator のマニュアルページを参照)
※本サンプルソースコードはnAG数値計算ライブラリ(Windows, Linux, MAC等に対応)の関数を呼び出します。
サンプルのコンパイル及び実行方法
| このソースコードをダウンロード |
/* nag_mv_cluster_indicator (g03ejc) Example Program.
*
* CLL6I261D/CLL6I261DL Version.
*
* Copyright 2017 Numerical Algorithms Group.
*
* Mark 26.1, 2017.
*
*/
#include <nag.h>
#include <stdio.h>
#include <nag_stdlib.h>
#include <nagg03.h>
#define X(I, J) x[(I) *tdx + J]
int main(void)
{
Integer exit_status = 0, i, *ic = 0, *ilc = 0, *iord = 0, *isx = 0;
Integer *iuc = 0;
Integer j, k, m, n, nsym, tdx;
NagError fail;
Nag_ClusterMethod method;
Nag_DistanceType dist;
Nag_MatUpdate update;
Nag_VarScaleType scale;
char nag_enum_arg[40];
char **c = 0, name[40][3];
double *cd = 0, *d = 0, dlevel, dmin_, *dord = 0, dstep, *s = 0;
double *x = 0, ydist;
INIT_FAIL(fail);
printf("nag_mv_cluster_indicator (g03ejc) Example Program Results\n\n");
/* Skip heading in data file */
scanf("%*[^\n]");
scanf("%ld", &n);
scanf("%ld", &m);
if (n >= 2 && m >= 1) {
if (!(cd = nAG_ALLOC(n - 1, double)) ||
!(d = nAG_ALLOC(n * (n - 1) / 2, double)) ||
!(dord = nAG_ALLOC(n, double)) ||
!(s = nAG_ALLOC(m, double)) ||
!(x = nAG_ALLOC((n) * (m), double)) ||
!(ic = nAG_ALLOC(n, Integer)) ||
!(ilc = nAG_ALLOC(n - 1, Integer)) ||
!(iord = nAG_ALLOC(n, Integer)) ||
!(isx = nAG_ALLOC(m, Integer)) || !(iuc = nAG_ALLOC(n - 1, Integer)))
{
printf("Allocation failure\n");
exit_status = -1;
goto END;
}
tdx = m;
}
else {
printf("Invalid n or m.\n");
exit_status = 1;
return exit_status;
}
scanf("%39s%*[^\n] ", nag_enum_arg);
/* nag_enum_name_to_value (x04nac).
* Converts nAG enum member name to value
*/
method = (Nag_ClusterMethod) nag_enum_name_to_value(nag_enum_arg);
scanf("%39s", nag_enum_arg);
update = (Nag_MatUpdate) nag_enum_name_to_value(nag_enum_arg);
scanf("%39s", nag_enum_arg);
dist = (Nag_DistanceType) nag_enum_name_to_value(nag_enum_arg);
scanf("%39s%*[^\n] ", nag_enum_arg);
scale = (Nag_VarScaleType) nag_enum_name_to_value(nag_enum_arg);
for (j = 0; j < n; ++j) {
for (i = 0; i < m; ++i)
scanf("%lf", &X(j, i));
scanf("%2s", name[j]);
}
for (i = 0; i < m; ++i)
scanf("%ld", &isx[i]);
for (i = 0; i < m; ++i)
scanf("%lf", &s[i]);
scanf("%ld", &k);
scanf("%lf", &dlevel);
/* Compute the distance matrix */
/* nag_mv_distance_mat (g03eac).
* Compute distance (dissimilarity) matrix
*/
nag_mv_distance_mat(update, dist, scale, n, m, x, tdx, isx, s, d, &fail);
if (fail.code != NE_NOERROR) {
printf("Error from nag_mv_distance_mat (g03eac).\n%s\n", fail.message);
exit_status = 1;
goto END;
}
/* Perform clustering */
/* nag_mv_hierar_cluster_analysis (g03ecc).
* Hierarchical cluster analysis
*/
nag_mv_hierar_cluster_analysis(method, n, d, ilc, iuc, cd, iord, dord,
&fail);
if (fail.code != NE_NOERROR) {
printf("Error from nag_mv_cluster_indicator (g03ejc).\n%s\n",
fail.message);
exit_status = 1;
goto END;
}
printf("\nDistance Clusters Joined\n\n");
for (i = 0; i < n - 1; ++i) {
printf("%10.3f ", cd[i]);
printf("%3s", name[ilc[i] - 1]);
printf("%3s", name[iuc[i] - 1]);
printf("\n");
}
/* Produce dendrogram */
nsym = 20;
dmin_ = 0.0;
dstep = cd[n - 2] / (double) nsym;
/* nag_mv_dendrogram (g03ehc).
* Construct dendrogram following
* nag_mv_hierar_cluster_analysis (g03ecc)
*/
nag_mv_dendrogram(Nag_DendSouth, n, dord, dmin_, dstep, nsym, &c, &fail);
if (fail.code != NE_NOERROR) {
printf("Error from nag_mv_dendrogram (g03ehc).\n%s\n", fail.message);
exit_status = 1;
goto END;
}
printf("\n");
printf("Dendrogram ");
printf("\n");
printf("\n");
ydist = cd[n - 2];
for (i = 0; i < nsym; ++i) {
if ((i + 1) % 3 == 1) {
printf("%10.3f%6s", ydist, "");
printf("%s", c[i]);
printf("\n");
}
else {
printf("%16s%s", "", c[i]);
printf("\n");
}
ydist -= dstep;
}
printf("\n");
printf("%14s", "");
for (i = 0; i < n; ++i) {
printf("%3s", name[iord[i] - 1]);
}
printf("\n");
/* nag_mv_dend_free (g03xzc).
* Frees memory allocated to the dendrogram array in
* nag_mv_dendrogram (g03ehc)
*/
nag_mv_dend_free(&c);
/* nag_mv_cluster_indicator (g03ejc).
* Construct clusters following
* nag_mv_hierar_cluster_analysis (g03ecc)
*/
nag_mv_cluster_indicator(n, cd, iord, dord, &k, &dlevel, ic, &fail);
if (fail.code != NE_NOERROR) {
printf("Error from nag_mv_cluster_indicator (g03ejc).\n%s\n",
fail.message);
exit_status = 1;
goto END;
}
printf("\n%s%2ld%s\n\n", "Allocation to ", k, " clusters");
printf("Object Cluster\n\n");
for (i = 0; i < n; ++i) {
printf("%5s%s%5s", "", name[i], "");
printf("%ld ", ic[i]);
printf("\n");
}
END:
nAG_FREE(cd);
nAG_FREE(d);
nAG_FREE(dord);
nAG_FREE(s);
nAG_FREE(x);
nAG_FREE(ic);
nAG_FREE(ilc);
nAG_FREE(iord);
nAG_FREE(isx);
nAG_FREE(iuc);
return exit_status;
}
