Keyword: 2項誤差, 一般化線形モデル, フィット
概要
本サンプルは2項誤差をもつ一般化線形モデルのフィットを行うC言語によるサンプルプログラムです。 本サンプルは以下に示されるデータについて一般化線形モデルのフィットを行います。
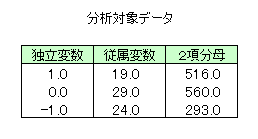
※本サンプルはnAG Cライブラリに含まれる関数 nag_glm_binomial() のExampleコードです。本サンプル及び関数の詳細情報は nag_glm_binomial のマニュアルページをご参照ください。
ご相談やお問い合わせはこちらまで
入力データ
(本関数の詳細はnag_glm_binomial のマニュアルページを参照)| このデータをダウンロード |
nag_glm_binomial (g02gbc) Example Program Data Nag_Logistic Nag_MeanInclude Nag_FALSE 3 1 0 1.0 19. 516. 0.0 29. 560. -1.0 24. 293. 1
- 1行目はタイトル行で読み飛ばされます。
- 2行目の1番目のパラメータ(link)はどのリンク関数が使用されるかを指定しています。 "Nag_Logistic"はログリンクが使用されることを意味します。2番目のパラメータは一般化線形モデルの引数に切片(mean)が含まれるかどうかを指定しています。 "Nag_MeanInclude" は切片を含めることを意味します。3番目のパラメータ(weight)は重みづけをするかどうかを指定します。 "Nag_FALSE" は重みづけをしないことを意味します。4番目のパラメータ(n)は観測値の数、5番目のパラメータ(m)は独立変数の数を指定しています。6番目のパラメータ(print_iter)は反復についての情報が必要かどうか、また出力する割合を指定しています。 "0"は何も出力しないことを意味します。
- 3〜5行目に独立変数の観測値(x)、従属変数の観測値(y)と2項分母(binom)を指定しています。
- 6行目はモデルに独立変数が含まれるかどうか(ivar(本関数の引数sx))を指定しています。"1"は含まれることを意味します。
出力結果
(本関数の詳細はnag_glm_binomial のマニュアルページを参照)| この出力例をダウンロード |
nag_glm_binomial (g02gbc) Example Program Results
Deviance = 7.3539e-02
Degrees of freedom = 1.0
Estimate Standard error
-2.8682 0.1217
-0.4264 0.1598
binom y fitted value Residual Leverage
516.0 19.0 18.45 0.1296 0.769
560.0 29.0 30.10 -0.2070 0.422
293.0 24.0 23.45 0.1178 0.809
- 3行目にデビアンス(deviance)が出力されています。
- 4行目に自由度が出力されています。
- 6〜16行目に一般化線形モデルの引数の推定値と標準誤差が出力されています。
- 18〜34行目に2項分母、従属変数の観測値、フィットされた値、残差、てこ値が出力されています。
ソースコード
(本関数の詳細はnag_glm_binomial のマニュアルページを参照)
※本サンプルソースコードはnAG数値計算ライブラリ(Windows, Linux, MAC等に対応)の関数を呼び出します。
サンプルのコンパイル及び実行方法
| このソースコードをダウンロード |
/* nag_glm_binomial (g02gbc) Example Program.
*
* CLL6I261D/CLL6I261DL Version.
*
* Copyright 2017 Numerical Algorithms Group.
*
* Mark 26.1, 2017.
*
*/
#include <nag.h>
#include <stdio.h>
#include <nag_stdlib.h>
#include <ctype.h>
#include <nagg02.h>
#define X(I, J) x[(I) *tdx + J]
#define V(I, J) v[(I) *tdv + J]
int main(void)
{
Integer exit_status = 0, i, *ivar = 0, j, m, max_iter, n, nvar, print_iter;
Integer rank, tdv, tdx;
Nag_IncludeMean mean;
Nag_Link link;
Nag_Boolean weight;
char nag_enum_arg[40];
double dev, df, eps, tol;
double *beta = 0, *binom = 0, *cov = 0, *offsetptr = 0, *se = 0;
double *v = 0, *wt = 0, *wtptr, *x = 0, *y = 0;
NagError fail;
INIT_FAIL(fail);
printf("nag_glm_binomial (g02gbc) Example Program Results\n");
/* Skip heading in data file */
scanf("%*[^\n]");
scanf("%39s", nag_enum_arg);
/* nag_enum_name_to_value (x04nac).
* Converts nAG enum member name to value
*/
link = (Nag_Link) nag_enum_name_to_value(nag_enum_arg);
scanf("%39s", nag_enum_arg);
mean = (Nag_IncludeMean) nag_enum_name_to_value(nag_enum_arg);
scanf("%39s", nag_enum_arg);
weight = (Nag_Boolean) nag_enum_name_to_value(nag_enum_arg);
scanf("%ld %ld %ld", &n, &m, &print_iter);
if (n >= 2 && m >= 1) {
if (!(binom = nAG_ALLOC(n, double)) ||
!(wt = nAG_ALLOC(n, double)) ||
!(x = nAG_ALLOC(n * m, double)) ||
!(y = nAG_ALLOC(n, double)) || !(ivar = nAG_ALLOC(m, Integer)))
{
printf("Allocation failure\n");
exit_status = -1;
goto END;
}
tdx = m;
}
else {
printf("Invalid n or m.\n");
exit_status = 1;
return exit_status;
}
if (weight) {
wtptr = wt;
for (i = 0; i < n; i++) {
for (j = 0; j < m; j++)
scanf("%lf", &X(i, j));
scanf("%lf%lf%lf", &y[i], &binom[i], &wt[i]);
}
}
else {
wtptr = (double *) 0;
for (i = 0; i < n; i++) {
for (j = 0; j < m; j++)
scanf("%lf", &X(i, j));
scanf("%lf%lf", &y[i], &binom[i]);
}
}
for (j = 0; j < m; j++)
scanf("%ld", &ivar[j]);
/* Calculate nvar */
nvar = 0;
for (i = 0; i < m; i++)
if (ivar[i] > 0)
nvar += 1;
if (mean == Nag_MeanInclude)
nvar += 1;
if (!(beta = nAG_ALLOC(nvar, double)) ||
!(v = nAG_ALLOC((n) * (nvar + 6), double)) ||
!(se = nAG_ALLOC(nvar, double)) ||
!(cov = nAG_ALLOC(nvar * (nvar + 1) / 2, double)))
{
printf("Allocation failure\n");
exit_status = -1;
goto END;
}
tdv = nvar + 6;
/* Set other control parameters */
max_iter = 10;
tol = 5e-5;
eps = 1e-6;
/* nag_glm_binomial (g02gbc).
* Fits a generalized linear model with binomial errors
*/
nag_glm_binomial(link, mean, n, x, tdx, m, ivar, nvar, y, binom, wtptr,
offsetptr, &dev, &df, beta, &rank,
se, cov, v, tdv, tol, max_iter, print_iter, "",
eps, &fail);
if (fail.code == NE_NOERROR || fail.code == NE_SVD_NOT_CONV ||
fail.code == NE_LSQ_ITER_NOT_CONV ||
fail.code == NE_RANK_CHANGED || fail.code == NE_ZERO_DOF_ERROR) {
if (fail.code != NE_NOERROR) {
printf("Error from nag_glm_binomial (g02gbc).\n%s\n", fail.message);
}
printf("\nDeviance = %13.4e\n", dev);
printf("Degrees of freedom = %3.1f\n\n", df);
printf(" Estimate Standard error\n\n");
for (i = 0; i < nvar; i++)
printf("%14.4f%14.4f\n", beta[i], se[i]);
printf("\n");
printf(" binom y fitted value Residual Leverage\n\n");
for (i = 0; i < n; ++i) {
printf("%10.1f%7.1f%10.2f%12.4f%10.3f\n", binom[i], y[i],
V(i, 1), V(i, 4), V(i, 5));
}
}
else {
printf("Error from nag_glm_binomial (g02gbc).\n%s\n", fail.message);
exit_status = 1;
goto END;
}
END:
nAG_FREE(binom);
nAG_FREE(wt);
nAG_FREE(x);
nAG_FREE(y);
nAG_FREE(beta);
nAG_FREE(v);
nAG_FREE(se);
nAG_FREE(cov);
nAG_FREE(ivar);
return exit_status;
}
